Edición de Genomas de Plantas
Octubre 29 de 2019
Alejandro Hernández, Director de Biotecnología para Centroamérica y el Caribe de CropLife Latin America
La edición de genomas en plantas se logra gracias a la biotecnología de precisión y apunta a ser un hito de la agricultura moderna. Actualmente hay solicitudes de permiso de venta en Estados Unidos de semillas de tomates, arroz, maíz, trigo, soya y champiñones. La edición genética promete cambios nutricionales como la producción de trigo libre de gluten o reducción de grasas trans en aceites de soya. ¿En qué consiste?
El genoma es la totalidad del material genético de un organismo o una especie en particular. En el caso de organismos superiores o eucariotas, como las plantas y los animales, existe el genoma nuclear y el genoma de los plastidios (plantas) y/o mitocondria (animales y plantas).
El material genético se presenta en estructuras altamente organizadas en el ADN, y es un código genético escrito en cuatro letras: A (adenina) que hace par con la T (timina), y la C (citosina) que hace par con G (guanina).

La secuencia de un genoma de plantas es similar a una biblioteca de letras. Dentro de esta librería de letras, se encuentra una pequeña proporción que corresponde a genes, o secuencias con alguna función. Estos genes tienen pequeños cambios que pueden variar su función. En mejoramiento genético convencional, estos cambios son las características que se buscan por polinización o por cambios azarosos. Hasta hace tan solo un par de décadas conocíamos muy poco de los genomas y aún menos de cómo ajustarlos, mejorarlos o repararlos cuando encontramos un error. Hoy, las herramientas de secuenciación y nuevas técnicas de Edición de Genomas hacen posible realizar y verificar con extrema precisión ajustes en el material genético. En el caso de las plantas, el escenario es muy prometedor ya que se puede obtener lo mismo que en mejoramiento convencional, pero en un tiempo mucho menor. Por esto, se les llaman nuevas técnicas de fitomejoramiento, en inglés New Breeding Techniques/ Technologies (NBTs).
Las tecnologías de edición de genomas se basan en 2 principios. El primero es que se debe ubicar el sitio preciso en el genoma para poder realizar alguna función. La más común, pero no la única, es la de cortar la doble hebra de ADN (en inglés “double-strand breaks” (DSBs)). El segundo principio es el uso de los mecanismos naturales de reparación del genoma. En organismos superiores o eucariotas se le llama reparación homóloga cuando se usa una copia idéntica de respaldo para reparar el genoma; o no homóloga cuando se repara el genoma sin la ayuda de una hebra de reparación. El resultado final es una secuencia de ADN con reparaciones o ajustes, a lo que en la literatura se les llamó originalmente Técnicas “SDN” o “SSN” del inglés “site directed nucleases”, o “site specific nucleases” 1, 2 y 3.

Exploremos estos dos conceptos para entender el estado del arte de las nuevas técnicas de edición de genomas.
Principio 1. Ubicar en el genoma el lugar correcto
Los primeros métodos para identificar algún material genético se identificaron en los 80s como mecanismos de defensa contra virus. Imaginen una tijeras de ADN, que en aquel entonces se llamaron endonucleasas y meganucleasas, éstas tenían la habilidad de reconocer alrededor de 6 a 8 letras las primeras y de 12-40 letras las segundas y realizar un corte en el ADN. En los 90s y principios del 2000 se avanzó con la generación de las nucleasas de dedos de Zinc (reconocen de 6-18pb) y las TALENS (Transcription-Activator-Like Effector Nucleases) que reconocen de 12-31 pares de bases. Mientras que, del 2012 a la fecha, se identifican una serie de tijeras o enzimas moleculares llamadas CAS asociadas a librerías de pequeños fragmentos de aproximadamente 20 pares de bases de secuencias de virus llamadas CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats). Estas últimas son las que se llaman CRISPR-CAS9/ CAS12a, CAS12b, CAS12e, CAS13a, CAS13b, CAS14.
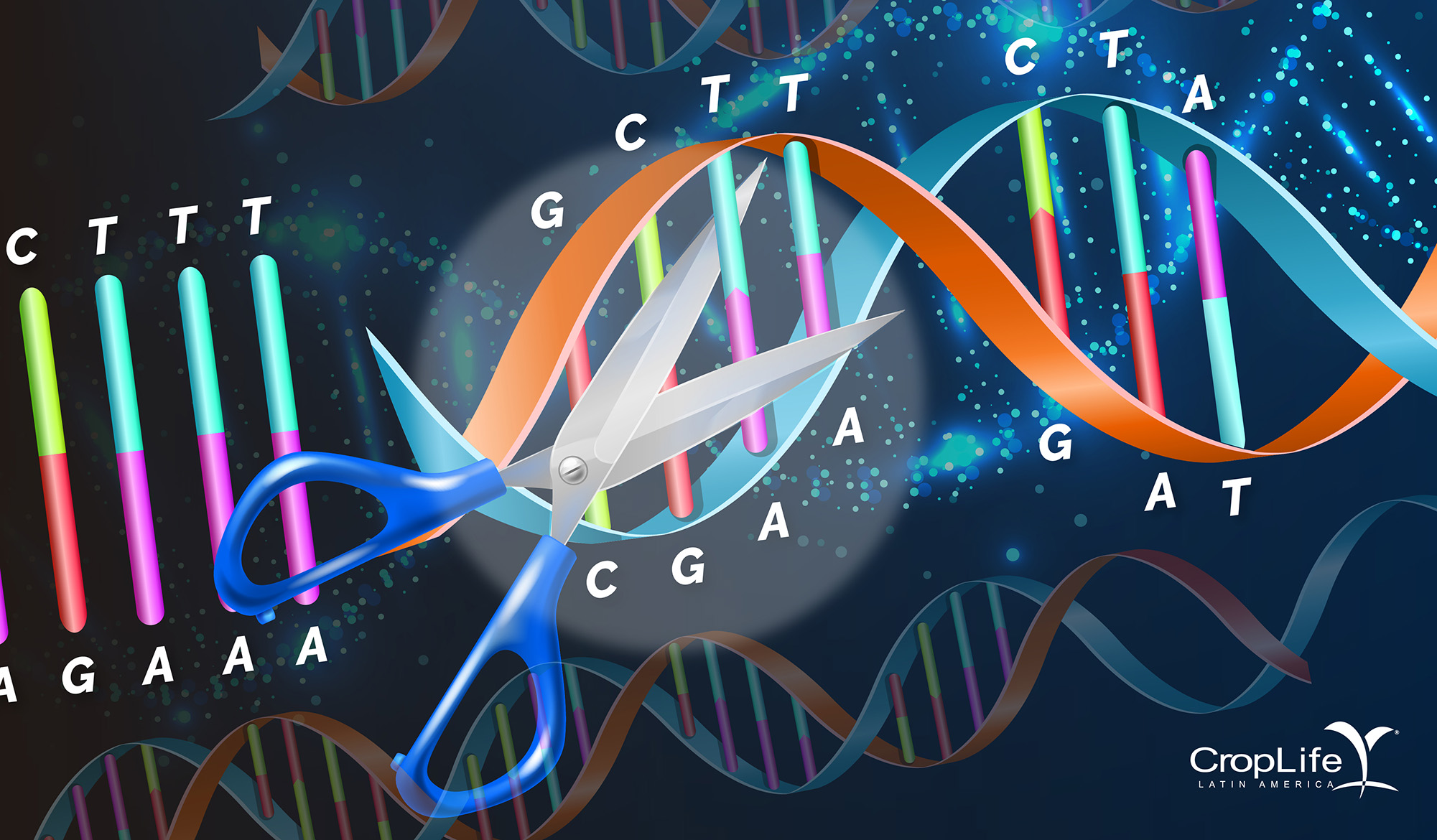
La historia de CRISPR-CAS9 es cautivadora
La enzima la descubrió Francisco Mojica en 1993 cuando estudió el microorganismo Haloferax mediterranei y notó un sistema de defensa en bacterias contra virus, conformado por dos elementos, una tijera que llamó CAS y una biblioteca de información que usaba la tijera para identificar el virus. A ésta última le denominó CRISPR en alusión a la seguidilla de secuencias de virus que se encontraban muy ordenadas una después de otra y separadas por un ADN idéntico que se repetía una y otra vez, de ahí el acrónimo “Clustered Regularly Interspaced Short Palindromic Repeats”.

El descubrimiento pasó por muchos años con un perfil bajo, hasta el 2012, cuando se verificó que el sistema bacteriano podía ser utilizado para editar ADN genómico. Es ahí cuando aparece en el escenario internacional una competencia científica al más alto nivel entre dos universidades en Estados Unidos. La Universidad de Berkeley en la costa oeste bajo el mando de las dos científicas Jennifer Doudna y Emmanuelle Charpentier1 y el instituto Broad de Harvard-MIT en la costa este liderado por el científico de Feng Zhang. El descubrimiento de ambos grupos se basó en que CAS9 (la tijera) se complementa perfectamente con una secuencia que podía diseñarse a la medida llamada ARNguía, que contiene 20 letras que usa de referencia para ubicar un lugar particular en el genoma, haciéndola sumamente específica.
Principio 2, reparación del genoma
Una vez que se realiza un corte en la doble hebra de ADN, la maquinaria de reparación natural de la célula entra en acción. La maquinaria celular puede reparar de distintas maneras el genoma, y según el tipo de reparación se le llama “SDN” o “SSN” del inglés “site directed nucleases”, o “site specific nucleases” 1, 2 y 3.
La SDN o SSN-1 son producto de la recombinación no homóloga, es decir es una reparación que no usa una hebra de reparación y resulta en inserciones, deleciones o pequeños cambios.
Las SDS-2 usan un ADN hebra y por tanto la reparación es homóloga, pero no insertan un ADN nuevo, sino que reparan el mensaje, por ejemplo, pueden ocasionar cambios a nivel de los aminoácidos de la proteína que codifica.
Las SDN-3 o SSN-3 usan una hebra de reparación, mediante recombinación homóloga y por tanto pueden resultar o no, en una inserción nueva de ADN. Esto es importante de entender dado que es posible insertar un alelo completo en una ubicación específica en el genoma. Si esta inserción es idéntica a lo que se logra por mejoramiento convencional, el resultado final es indistinguible de las técnicas convencionales.
Nos encontramos en una época fascinante, donde convergen distintas tecnologías hacia una agricultura sostenible. Sin lugar a duda, la biotecnología de precisión jugará un papel clave en producir más y de manera más equilibrada.
Bibliografía
1. Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., & Charpentier, E. (2012). A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity. science, 337(6096), 816-821.















